 Forschende der Uni Graz berechnen mit KI die Oberflächen-Strukturen von Bakterien, Archaeen und Viren, die sich mit einer Hülle aus Proteinen vor Angriffen von außen schützen. Bei diesen Oberflächen handelt es sich um komplexe regelmäßige, symmetrische Strukturen. SymProFold kann von allen Wissenschaftler:innen für Forschungszwecke genutzt werden. Das Forschungsteam: , Christoph Grininger, Theo Sagmeister, Tea Pavkov-Keller, Christoph Buhlheller und Nina Gubensäk (v.l.). Foto: (c) Uni Graz/Tzivanopoulos
Forschende der Uni Graz berechnen mit KI die Oberflächen-Strukturen von Bakterien, Archaeen und Viren, die sich mit einer Hülle aus Proteinen vor Angriffen von außen schützen. Bei diesen Oberflächen handelt es sich um komplexe regelmäßige, symmetrische Strukturen. SymProFold kann von allen Wissenschaftler:innen für Forschungszwecke genutzt werden. Das Forschungsteam: , Christoph Grininger, Theo Sagmeister, Tea Pavkov-Keller, Christoph Buhlheller und Nina Gubensäk (v.l.). Foto: (c) Uni Graz/Tzivanopoulos
Ein Team um Tea Pavkov-Keller an der Universität Graz hat eine Methode entwickelt, mit der sich die Struktur der Hülle von Bakterien, Archaeen und Viren mit Hilfe künstlicher Intelligenz zuverlässig bis ins kleinste Detail berechnen lässt. Das spart enorm viel Zeit und Aufwand im Labor. Im Fachmagazin Nature Communications wird das neue Verfahren mit dem Namen „SymProFold“ erstmals vorgestellt.
Zielgerichtete Experimente durch errechnetes Vorwissen
Wofür Forscher:innen im Labor bisher Monate oder gar Jahre gebraucht haben, das lässt sich nun am Computer in wenigen Stunden berechnen. „Wir haben mit SymProFold eine Methode entwickelt und optimiert, mit der wir in kurzer Zeit die Struktur von komplexen regelmäßigen Protein-Assemblierungen berechnen können“, berichten Christoph Buhlheller und Theo Sagmeister vom Institut für Molekulare Biowissenschaften der Uni Graz. Alles, was Forscher:innen dazu brauchen, ist eine Sequenz des Proteins, aus dem das Gitter aufgebaut ist. Die Bestätigung der errechneten Struktur im Labor sichert das Ergebnis ab. Durch das rechnerisch erworbene Vorwissen können die Experimente zielgerichtet durchgeführt werden, wodurch wertvolle Zeit eingespart wird.
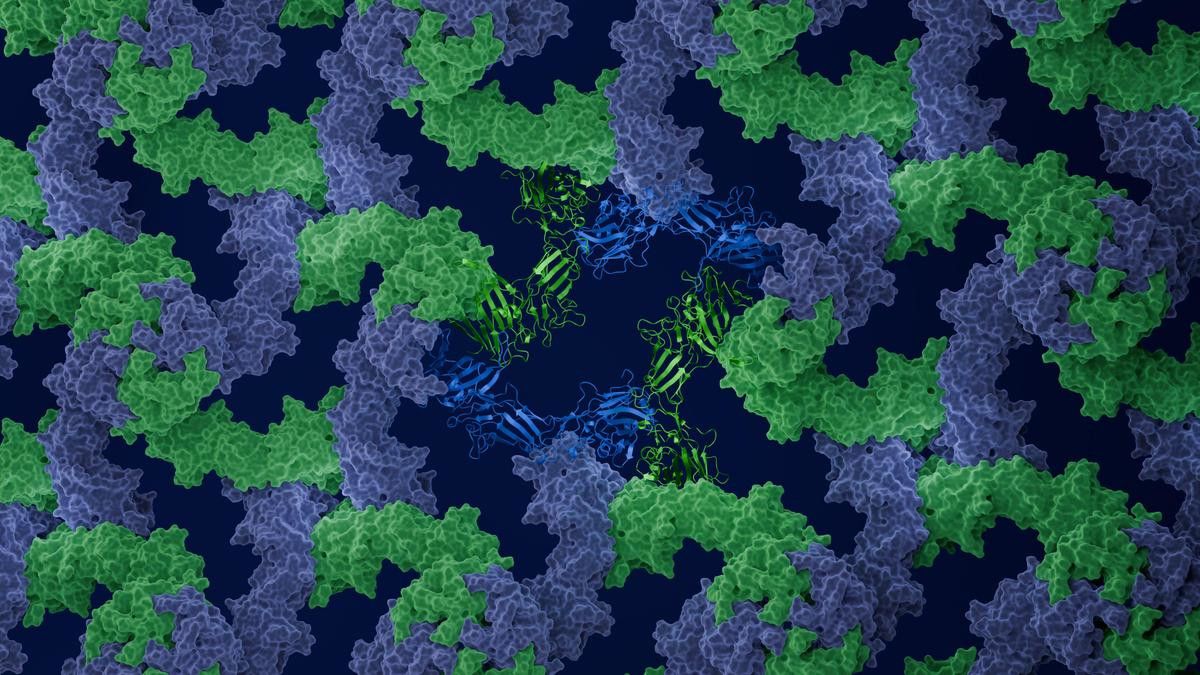
Das Bild zeigt den S-Layer von Aeromonas salmonicida, berechnet mit SymProFold. Die einzelnen Proteinuntereinheiten (blau/grün) assemblieren zu dem symmetrisch aufgebauten S-Layer. (c) Uni Graz/Tzivanopoulos
Schlüssel zum Nano-Design für Medizin und Technik
Jede Art von Virus, Archaea oder Bakterium hat eine eigene Oberflächenstruktur. Virenhüllen werden Capside genannt, jene von Bakterien und Archaea S-Layer – S für surface, deutsch Oberfläche. Letztere sind besonders komplexe, supramolekulare Gebilde. „Wenn wir genau wissen, wie diese Strukturen aussehen, kennen wir auch ihre Stärken und Schwächen“, erklärt Tea Pavkov-Keller, Leiterin der Arbeitsgruppe. „Jene Stellen, an denen die Interaktionen stattfinden, können Angriffspunkte für Medikamente sein. Denn wenn Wirkstoffe dort Interaktionen verhindern, kann sich die Schutz-Hülle nicht mehr bilden“, führt die Wissenschaftlerin aus.
Dieses Wissen ist auch für andere Bereiche des Nano-Designs nützlich. S-Layer sind für die Entwicklung verschiedenster Anwendungen interessant, etwa Biosensoren oder Beschichtungen. Die genaue Kenntnis der Protein-Gitter-Struktur erlaubt es, ihnen durch gezielte Veränderungen ganz bestimmte Eigenschaften zu verleihen.
SymProFold kann von allen Wissenschaftler:innen für Forschungszwecke genutzt werden.
Die Forschungen sind Teil des Profilbereichs BioHealth der Universität Graz.
https://biohealth.uni-graz.at
Publikation:
SymProFold: Structural prediction of symmetrical biological assemblies
Christoph Buhlheller, Theo Sagmeister, Christoph Grininger, Nina Gubensäk, Uwe B. Sleytr, Isabel Usón, Tea Pavkov-Keller
Nature Communications, 18. 9. 2024
https://doi.org/10.1038/s41467-024-52138-3 (https://www.nature.com/articles/s41467-024-52138-3)
Kontakt:
Ass.-Prof. Dr. Tea Pavkov-Keller
Institut für Molekulare Biowissenschaften
Universität Graz
Tel.: +43 316 380 5483
E-Mail: tea.pavkov@uni-graz.at. September
(GZ)
Quelle: Universität Graz
Foto: (C)Uni Graz/Tzivanopoulos
Bei Rückfragen zum Artikel wenden Sie sich
bitte direkt an die im Artikel angegebene Kontaktadresse.






